لغت نامه مهندسی ژنتیک برگرفته از کتاب کلون سازی ژن ها و آنالیز DNA
سایت ویستاژن لغت نامه مهندسی ژنتیک از کتاب کلون سازی ژن ها و آنالیز DNA نوشته پروفسور براون را در اختیار شما عزیزان قرار داده است.
برای راحت پیدا کردن کلمه مورد نظرتان می توانید: دکمه اف و کنترل را هم زمان فشار دهید ( F و Ctrl) و در باکس باز شده لغت مورد نظرتان را سرچ کنید.
آدنوویروس : (Adenovirus) : ویروس جانوری که از مشتقات آن برای کلون کردن ژن ها در سلول های پستانداران استفاده می شود .
آرایش فضایی : (Conformation) : سازمانیابی فضایی یک مولکول . اشکال خطی و حلقوی ، آرایشهای فضایی ممکن برای پلینوکلئوتیدها هستند .
آگروباکتریوم تومه فاشینس : (Agrobacterium tumefaciens) : باکتری خاکزی که اگر دارای پلاسمید Ti باشد میتواند در تعدادی از گونههای گیاهان دو لپهای ایجاد گال تاجی کند .
آنتی سنس RNA:
آنالیز پیوستگی RFLP :
(RFLP Linkage analysis) : روشی است که از RFLP به عنوان نشانگری برای حضور یک الل خاص در نمونه DNA استفاده میکند . اغلب از این روش برای غربالگری افرادی که حامل ژن معیوب عامل یک بیماری ژنتیکی هستند ، استفاده میشود .
آنالیز حذف : ( Deletion analysis) : شناسایی توالیهای کنترل کننده یک ژن با مشخص کردن اثر حذف ناحیه خاصی از فرادست ژن بر بیان آن .
آنالیز با آنزیم های محدودگر : (Restriction analysis) : تعیین تعداد و اندازه قطعات DNA حاصل از برش یک قطعه DNA با یک آنزیم محدودگر.
آنزیم های محدودگر : (Restriction endonuclease) : اندنوکلئازهایی هستند که مولکولهای DNA را تنها در توالیهای نوکلئوتیدی خاص میبرند .
آنیلینگ : (Annealing) : اتصال یک الیگونوکلئوتید به یک مولکول DNA تک رشتهآی از طریق هیبرید شدن با آن .
ابرمارپیچ : (Supercoiled) : آرایش فضایی DNA حلقوی که در اثر فشار کششی به شکلی شبیه سیم تلفن در میآید .
اِپی زوم : (Episome) : پلاسمیدی که می-تواند درون کروموزوم سلول میزبان وارد شود .
اتورادیوگرافی : (Autoradiography) : روشی برای ظاهر سازی مولکولهایی که با مواد رادیواکتیو نشاندار شدهاند. در این روش از یک فیلم عکاسی حساس به اشعه X استفاده میشود .
اتیدیوم برماید : (Ethidium bromide) : یک ماده شیمیایی فلورسنت که میان جفت بازهای مولکول DNA دورشتهای وارد میشود و از آن برای تشخیص DNA استفاده میشود .
اثر جایگاهی : (Positional effect) : تفاوت در میزان بیان ژنهایی که در جایگاههای مختلفی از ژنوم کلون شدهاند .
ارزیابی تداخل ناشی از تغییر : ( Modification interference assay) : روشی است که از تغییرات شیمیایی برای مشخص کردن نوکلئوتیدهای درگیر در برهم کنشهای DNA با پروتئین متصل شونده به آن سود میجوید.
اِسفروپلاست : (Sphaeroplast) : سلولی که بخشهایی که از دیواره سلولیاش را از دست داده است .
اضافه کردن ژن : (Gene addition ) : از استراتژیهای مهندسی ژنتیک که شامل وارد کردن یک یا چند ژن جدید به درون یک موجود زنده است .
اگزوتروف : (Auxotroph) : میکروارگانیسم جهش یافتهای که تنها در حضور ماده مغذی رشد میکند که سویه وحشی از آن بینیاز است .
اگزونوکلئاز : (Exonuclease) : آنزیمی که نوکلئوتیدها را تکتک از یک انتهای مولکول اسید نوکلئیک جدا میکند .
القا : (Induction) : (1) در مورد یک ژن : روشن شدن بیان یک ژن یا چند ژن در پاسخ به حضور یک ماده شیمیایی یا تحریکات دیگر
(2) در مورد فاژ λ : خارج شدن ژنوم λ وارد شده به درون ژنوم باکتری و ورود آن به فاز لیتیک در پاسخ به یک ماده شیمیایی یا تحریکات دیگر .
الکتروپوریشن : (Electroporation ) : روشی برای افزایش جذب DNA توسط پروتوپلاستها از طریق اعمال ولتاژ بالا که موجب ایجاد سوراخهای ریز موقت در غشای سلول میشود .
الکتروفورز : (Electrophoresis) : جدا سازی مولکولها بر اساس نسبت بار به جرم آنها .
الکتروفورز با میدان الکتریکی متغیر عمود بر هم : (Orthogonal field altetnation gel electrophoresis (OFAGE)) : روش الکتروفورز روی ژل که از میدان الکتریکی نوساندار برای جداسازی قطعات بزرگ DNA استفاده میشود .
الگو : (Template) : پلینوکلئوتیدها ( یا بخشی از یک پلینوکلئوتید) که ساخته شدن یک پلی نوکلئوتید مکمل را هدایت میکند .
المثنی سازی : (Replica Plating) روشی که کلونیها رشد کرده روی پلیت آگاردار به پلیت جدیدی منتقل میشوند ، به طوری که کلونیها جایگاه خود را در پلیت جدید نیز به همان ترتیبی که روی پلیت قبلی رشد کرده بودند ، حفظ میکنند .
الیگونوکلئوتید : (Oligonucleotide) : مولکول DNA کوچک ، مصنوعی و تک رشتهای مثل آنهایی که به عنوان پرایمر در PCR یا توالییابی DNA استفادهمیشوند .
انتخاب : (Selection) : جدا کردن کلونی که حامل مولکول DNA نوترکیب مورد نظر است .
انتخاب Lac :
(Lac selection) : روشی برای مشخص کردن باکتریهایی نوترکیبی که دارای حاملهایی هستند که ژن Lacz’ را دارند . باکتریها را روی محیط کشتی که دارای آنالوگ لاکتوز است ، کشت میدهند که در صورت وجود فعالیت β- گالاکتوزیدازی ، رنگ آبی ایجاد میشود .
انتقال ساترن : (Southern transfer) : روشی برای انتقال نوارهای DNA از ژل آگارز به غشای نایلونی یا نیتروسلولزی .
انتقال مستقیم ژن : (Direct gene transfer) : فرایند کلون کردن بدون استفاده از حامل ، کهژن به درون کروموزوم وارد میشود و میتواند درون میزبان همانند سازی کند .
انتقال نورترن : (Northern transfer) : روشی برای انتقال نوارهای RNA از ژل /اگارز به غشای نایلونی یا نیتروسلولزی .
انتقال وسترن : (Western transfer) : روشی برای انتقال نوارهای پروتئینی از ژل الکتروفورز به غشای پایه.
انتقال هسته : (Nuclear transfer) : روشی برای ایجاد جانوران ترانس ژنیک که شامل انتقال هسته یک سلول سوماتیک به درون تخمکی است که هسته آن را قبلا درآوردهاند .
انتهای چسبنده : (Sticky end) : انتهای یک مولکول DNA دورشتهای که یک بخش تک رشتهای دارد .
انتهای تراز : (Blunt end or flush end ) : انتهای DNA که بخش تک رشتهای ندارد .
انتهای ‘ 3 3′ terminua): ) : یکی از دو انتهای پلی نوکلئوتیدها که عبارت از گروه هیدروکسیل متصل به کربن ‘ 3 قند است .
انتهای ‘ 5 : ( 5′ terminus ) : یکی از دو انتهای پلی نوکلئوتیدها که عبارت از گروه فسفات متصل به کربن ‘ 5 قند است .
اندونوکلئاز : ( Endonuclease) : آنزیمی که پیوندی فسفودی استری درون یک مولکول لسید نوکلئیک را میشکند .
انکلوزیون بادی ها : ( Inclusion body) : رسوبات بلوری یا نیمه بلوری درون سلول که اغلب حاوی مقادیر بالایی از پروتئینهای نا محلول هستند .
انگشت نگاری ژنتیکی : (Gene fingerprinting ) : روش هیبریداسیونی که توزیع ژنومی یک توالی تکراری بسیار متغیر پراکنده را مشخص میکند . الگوی نوارهای ایجاد شده در این روش برای هر شخص کاملا اختصاصی است .
انگشت نگاری کلون : ( Clone fingerprinting) : روشی که در آن قطعات ِdna کلون شده را برای مشخص کردن آنهایی که با هم ، همپوشانی دارند ، مقایسه میکند .
اَویدین :(Avidin) : پروتئینی که تمایل بالایی به بیوتین دارد و در سیستمهایی که بر مبنای پروبهای بیوتیندار هستند استفاده میشود .
باکتریوفاژ یا فاژ : (Bacteriophage or Phage) : ویروسی که میزبانش باکتری است . مولکولهای DNA باکتریوفاژها اغلب به عنوان حاملهای کلون کردن استفاده میشود .
باکلوویروس : (Baculovirus) : ویروسی که به عنوان حامل کلون کردن برای تولید پروتئنهای نوترکیب در سلولهای حشرات استفاده میشود .
برچسب توالی بیان شونده : (Expressed sequence tag (EST) ) : توالی کامل یا ناقص cDNA
بُرزنی چندژنی : (Multigene shuffling) : راهکاری برای تکامل هدایت شده که عبارت است از انتخاب بخشهایی از هر یک از اعضای یک خانواده چندژنی و دوباره سر هم کردن بخشهایی از آنها برای ایجاد ژن جدید.
بسته بندی آزمایشگاهی : (In vitro packaging ) : ساخته شدن ذرات λعفونتزا با تولید پروتئینهای کپسید λ و زنجیری از مولکولهای DNA که میان آنها جایگاههای cos قرار گرفته است .
بلاست : (BLAST) : الگوریتمی که برای جستجوی شباهتهای توالیهای اسیدهای نوکلئیک یا پروتئینها استفاده میشود .
بیوانفورماتیک : (Bioinformatics) : استفاده از روشهای کامپیوتری برای مطالع ژنوم .
بیوتکنولوژی : (Biotechnology) : استفاده از فرایندهای بیولوژیک در صنعت و تکنولوژی (فن آوری) .
بیوتین : ( Biotin) : مولکولی که امکان متصل کردن آن به dUTP وجود دارد . به این ترتیب می توان پروب های DNA نشاندار شدهای به دست آورد که رادیواکتیو نیستند .
بیولیستیک : (Biolistics) : وسیلهای برای انتقال DNA به درون سلول ها . در این روش از بمباران سلول ها با پرتابه های میکروسکوپی آغشته به DNA استفاده می شود.
پایپلوما ویروسها : (Papillomaviruses) : گروهی از ویروس های پستانداران که از مشتقات آن به عنوان حامل های کلون کردن استفاده می شود .
پراکسیداز ترب : (Horseradish Peroxidase) : آنزیمی است که می تواند با DNA کمپلکس تشکیل دهد . از آن برای نشاندار کردن غیر رادیواکتیو DNA استفاده می شود .
پرایمر : (Primer) : الیگونوکلئوتید تک رشته ای کوتاهی که هرگاه از طریق جفت کردن بازهایش به مولکول تک رشته ای بچسبد، به عنوان نقطه شروعی برای ساخته شدن رشته مکمل توسط DNA پلیمراز عمل می کند .
پذیرا ( مستعد ) : (Competent) : کشت باکتریایی تیمار شده ای که توان دریافت DNA آن بالا رفته است .
پرکردن انتها : (End filling) : تبدیل انتهای چسبنده به انتهای تراز با آنزیمی که رشته مکمل ناحیه تک رشته ای را می سازد .
پروی هیبریداسیون : (Hybridization probe) : مولکول اسید نوکلئک نشانداری که می توان از آن برای شناسایی مولکول های مکمل یا همولوگ از طریق ایجاد هیبرید پایدار میان جفت بازها استفاده کرد .
پرتوپلاست : (Protoplast) : سلولی که دیواره سلولی آن کاملا برداشته شده است.
پروتئاز : (Protease) : آنزیمی که پروتئین ها را هضم می کند
پروتئوم : (Proteome) : محتوای پروتئینی یک سلول یا بافت.
پروتئومیکس : (Proteomics) : مجموعه روشهایی برای مطالعه پروتئوم.
پروتئین A : (Protein A ) : یکی از پروتئینهای باکتری استافیلوکوکوس اورئوس که به طور اختصاصی به مولکول های ایمونوگلوبین G ( که یک آنتی بادی است ) متصل می شود.
پروتئین نوترکیب : (Recombinant protein) : پلیپپتیدی که به عنوان محصول بیان یک ژن کلون شده در یک سلول نوترکیب تولید می شود.
پروفاژ : (Prophage) : شکل ادغام شده مولکول DNA یا فاژ لیزوژن را گویند.
پروفایلینگ DNA : (DNA profiling ) : یک روش PCR که الل های موجود در لوکوس های متفاوت STR در درون ژنوم را تعیین می کند، به این منظور که از اطلاعات DNA برای شناسایی افراد استفاده شود.
پروموتر : (Promoter) : توالی نوکلئوتیدی فرادست ژن که به عنوان نشانه ای برای اتصال RNA پلیمراز عمل می کند.
پروموتر ضعیف : (Weak promoter) : پروموتری با کارآیی اندک که ساخته شدن رونوشت های RNA را در مقادیر اندک امکان پذیر می سازد.
پروموتر قوی : (Strong promoter) : پروموتر کارآمدی که با سرعت بالایی رونویسی شده و ساخته شدن RNA را هدایت می کند.
پساژنومیک : (Post-genomics) : مطالعاتی ه برای شناسایی تمام ژن های ژنوم و عملکرد آن ها انجام می شود.
پلاسمید : (Plasmid) : قطعه DNA حلقوی که مستقل از کروموزوم میزبان است و اغلب در باکتری ها و گاه در دیگر انواع سلول ها مشاهده می شود.
پلاسمید 2 میکرومتری : (2ᶣm plasmid) : پلاسمیدی که بطور طبیعی در مخمر ساکارومیسس سرویزیه وجود دارد و به عنوان پایه ای برای مجموعه ای از حامل های کلون سازی بکار رفته است.
پلاسمید اپیزومال مخمر : (Yeast episomal plasmid (Yep)) : حامل کلون سازی مخمری که مبدا شروع همانندسازی کروموزوم 2 میکرومتری مخمر را دارد .
پلاسمید ادغام شونده مخمر : (Yeast integrative plasmid (Yip) ) : حامل مخمری که برای تکثیر ، وارد کروموزوم میزبان می شود.
پلاسمید تکثیر شونده مخمر : (Yeast replicative plasmid (YRp)) : حامل مخمری که دارای مبدا همانندسازی کروموزومی است.
پلاسمید چند نسخهای : (Multicopy plasmid ) : پلاسمیدس که با تعداد نسخه های بالایی در سلول وجود دارد.
پلاسمید غیرمسلح : (Disarmed plasmid) : پلاسمید Ti که برخی یا تمام ژن های T-DNA از آن خارج شده است. بنابراین نمی تواند موجب رشد سرطانی سلول های گیاهی شود.
پلاسمید وسیع الطیف : (Broad host range plasmid) : پلاسمیدی که می تواند در انواع متنوعی از میزبان ها تکثیر شود.
پلاسمید Ri : (Ri plasmid) : پلاسمید آگروباکتریوم ریزوژنز که شبیه پلاسمید Ti است و برای کلون کردن ژن در گیاهان عالی تر استفاده می شود.
پلاسمید : Ti ( Ti plasmid) : پلاسمید بزرگی که در سلولهای آگروباکتریوم تومهفاشینس وجود دارد و می تواند گل ناجی شکل را در تعدادی از گونه های گیاهی ایجاد کند .
پلاک : (Plaque) : ترکیب پلیمری که برای رسوب گذاری ماکرو مولکول ها و تجمعات مولکولی استفاده می شود
پلیاتیلیلن گلیکول : (polyethylene glycol) : ترکیب پلیمری که برای رسوب گذاری ماکرو مولکول ها و تجمعات مولکولی استفاده می شود.
پلیلینکر : (Polylinker) : الیگو نوکلئوتید دو رشته ای صناعی که چندین جایگاه برش برای آنزیم های محدودگر دارد .
پیلی : (Pilus) : از ساختارهای سطحی باکتری های حاوی پلاسمید کونژوگه شونده است و به نظر می رسد که DNA از طریق آن عبور می کند.
تاخیر حرکت در ژل : (Gel retardation ): روشی که قطعه DNA متصل شده به یک پرونئین را با کاهش میزان حرکت آن در ژل الکتروفورز مشخص می کند.
ناکاوت ژن : (Gen knockout) : روشی است که موجب غیر فعال شدن یک ژن می شود. از این روش برای مشخص کردن عملکرد یک ژن استفاده می شود .
تحلیل پیوستگی : (Linking analysis) : روشی است برای نقشه برداری جایگاه کروموزومی یک ژن با مقایسه الگوی توارثی آن با ژن ها یا جایگاه هایی که موقعیت آن ها روی کروموزوم مشخص است.
تحلیل رو نوشت : (Transcript analysis) : آزمایش هایی برای مشخص کردن بخشی از مولکول DNA که به RNA رونویسی شده اند.
تحلیل شجره نامه : (Pedigree analysis) : استفاده از شجره نامه خانوادگی انسان برای تحلیل وراثت یک نشانگر DNA
تحلیل فامیلی : (Kinship analysis ) : بررسی الگوی DNA یا انجام مطالعات دیگر برای مشخص کردن روابط فامیلی دو نفر.
تراشه DNA : (DNA chip) : تراشه سیلیکونی که تعداد زیادی آرایه الیگونوکلئوتیدهای مورد استفاده در مطالع ترانسکریپتوم و … را روی آن سوار کرده اند.
ترانسپوزون : (Transposon) : توالی DNA که می تواند از یک قسکت به قسمت دیگری از ژنوم برود.
ترانس ژنتیک : ( Transgnic) : حیوان یا گیاهی که یک ژن کلون شده را در تمام سلولهایش دارد.
ترانسفکت کردن : (Transfection) : وارد کردن مولکول های DNA ویروسی خالص شده به سلول زنده.
ترانسفورم کردن : (Transformation) : وارد کردن مولکول DNA به درون سلول زنده.
ترجمه با آزاد شدن از هیبرید ( Hybrid-release translation (HRT)) : روشی برای شناسایی پلیپپتیدی که توسط ژن کلون شده کد می شود.
ترجمه با حبس در هیبرید : ( Hybrid-arrest translation (HART)) : روشی برای مشخص کردن پلیپپتیدی که توسط یک ژن کلون شده کد می شود.
ترمیناتور : (Terminator) : توالی نوکلئوتیدی کوتاهی در فرودست ژن که به منزله علامت پایان رونویسی عمل می کند.
ترانسکریپتوم : (Transcriptome) : محتوی mRNA سلول یا بافت .
تعداد نسخه : (Copy number) : تعدادمولکول های یک نوع پلاسمید که در یک سلول حضور دارند.
تکامل چند ناحیهای : (Multiregional evolution) : تئوری که بر اساس آن انسان مدرن از جمعیتی از Homo erectus که در حدود یک میلیون سال پیش آفریقا را ترک کرده اند منشا گرفته است.
تکامل هدایت شده : (Direct evolution) : مجموعه ای از روش های آزمایشگاهی برای بدست آوردم ژن های جدید با محصولات بهتر.
تکثیر پلاسمید : ( Plasmid amplification) : روش گرماگذاری میکروب در حضور یک مهار کننده سنتز پروتئین ها، با هدف افزایش تعداد پلاسمیدهای حاضر در یک کشت باکتریایی.
تمایل کدونی : (Codon bias) : پدیدهای که موجب می شود کدون ها یک آمینواسید به میزان مساوی در ژن های یک موجود زنده استفاده نشوند.
توالی برچسب خورده : (Sequence tagged site (STS)) : توالی DNA که جایگاه آن رئی ژنوم نقشه برداری شده است .
توالی تکراری کوتاه پشت سرهم : ( Short tandem repeat (STR)) : چند شکلی نسخه های پشت سر هم از واحدهای دو، سه ، چهار یا پنج نوکلئوتیدی را شامل می شود. این توالی ها ریز ماهواره هم نامیده می شود.
توالی یابی با چرخه حرارتی : ( Thermal cycle sequencing) : روش توالی یابی DNA که از PCR برای ایجاد پلینوکلئوتیدهای خاتمه زنجیره استفاده می کند.
توالی توافقی : (Consensus sequence) : توالی نوکلئوتیدی مورد توافق برای توصیف نواحی خاصی از ژنوم . هر یک از جایگاه های توالی توافقی، نوکلئوتیدی را که اغلب در آن جایگاه دیده می شود نشان می دهد.
توالی تکراری بسیار متغیر پراکنده : ( Hypervariable dispersed repetitive sequence) : از توالی های تکراری DNA انسان که در انگشت نگاری DNA استفاده می شود.
توالییابی DNA : (DNA sequenceing ) : مشخص کردن ترتیب نوکلئوتیدها در مولکول DNA .
تئوری خارج از آفریقا : (Out of Africa hypothesis) : تئوری که بر اساس آن انسان مدرن در آفریقا تکامل یافته و در حدود 50 تا 100 هزار سال قبل در تمام جهان پراکنده شده و جایگزین اخلاف Homo ercuts شده است .
جایگاه اتصال ریبوزوم : (Ribosome binding site) : توالی نوکلئوتیدی کوتاهی در فرادست ژن است که بعد از رونویسی ، جایگاه اتصال ریبوزوم را تشکیل میدهد .
جانور ترانسژن : (Transgenic animal) : جانوری که یک ژن کلون شده را در تمام سلول هایش دارد.
جایگاه COS : ( COS site) : یکی از جایگاه های تک رشته ای و چسبنده حاضر در انتهاهای مولکول های DNA سویه خاصی از فاژ λ .
جزایر CPG : (CpG island ) : مناطقی از DNA که غنی از توالی CG هستند و در فرادست حدود 56% از ژن های انسان وجود دارند .
جستجوی همولوژی : (Homology search) : روشی که در /ان ژن هایی که توالی هایی مشابه ژن ناشناخته را دارند ، بررسی می شوند تا شناسایی ژن را تایید کنند یا عملکرد ژن ناشناخته را مشخص سازند .
جمع آوری : (Harvesting) : جدا کردن میکرو ارگانیسم ها از یک محیط کشت که معمولا با سانتریفوژ انجام می شود .
جِمینی ویروس : (Geminivirus) : یکی از دو گروه DNA ویروس هایی که گیاهان را عفونی می کنند . اعضای این گروه پتانسیل استفاده شدن به عنوان وکتورهای کلونسازی در برخی از گونه های گیاهان عالی را دارند .
جهش زایی آزمایشگاهی : (In vitro mutagenesis) : روشی برای ایجاد جهش خاص در جایگاه مشخصی از مولکول DNA.
جهش زایی با واسطه الیگو نوکلئوتیدها : (Oligonucleotide-directed mutagenesis) : روش جهش زایی آزمایشگاهی که شامل استفاده از الیگو نوکلئوتیدهای صناعی برای ایجاد تغییر نوکلئوتیدی مورد نظر در ژن است.
جهش حساس به حرارت : (Temperature-sensitive mutation ) : جهشی که باعث می شود محصول ژن در دامنه دمایی مهینی فعال باشد ( مثلا زیر C ͦ 30 ) اما در دماهای دیگر غیر فعال شود ( مثلا بالای C ͦ 30 ).
حامل وارد شوند : ( Insertion vector ) : حامل λ که بخشی از DNA غیر ضروری آن حذف شده است.
چارچوب خواندن باز : (ORF) (Open reading frame) : مجموعه کدون هایی که می توانند به همراه هم یک ژن را تشکیل دهند.
چارچوب خواندن : (Reading frame) : یکی از شش توالی همپوشان کدون های سه تایی را می گویند. روی هر رشته پلینوکلئوتید DNA دو رشته ای، سه قاب خواندن وجود دارد.
چرخه عفونی لیتیک : ( Lytic infection cycle ) : چرخ های که فاژ ، تکثیر یافته و بلافاصله بعد از شروع عفونت منجر به مرگ سلول میزبان می شود. ورود DNA فاژ به درون کروموزوم باکتری در این حالت رخ نمی دهد .
چرخه عفونی لیزوژنی : (Lysogenic infection cycle ) : چرخ های از عفونت فاژی که با وارد شدن DNA فاژ به درون کروموزوم میزبان همراه است.
چگالی شناوری : (Buoyant density) : چگالی یک مولکول یا ذره در هنگامی که درون یک محلول نمکی معلق شده است.
چند شکلی : (Polymorphism) : جایگاه ژنی که به صورت الل های مختلف با دیگر تفاوت ها در جمعیت دیده می شود.
چند شکلی اندازه قطعات حاصل از برش با آنزیم های محدودگر : (Restriction fragment length polymorphism (RFLP)) : جهشی است که موجب تغییر در جایگاه برش آنزیم محدودگر می شود و بنابراین الگوی حاصل از برش مولکول DNA توسط آن آنزیم محدودگر تغییر می یابد .
چند شکلی تک نوکلئوتیدی : ( Single nucleotide polymorphism (SNP)) : جهش نقطه ای که در برخی افراد جمعیت مشاهده می شود.
حامل جایگزینی : ( Replacement vector) : حاملλ که برای وارد کردن یک قطعه DNA طراحی شده و در آن DNA مورد نظر با یک یاز بخش های غیر ضروری مولکول DNA فاژ جایگزین شده است .
حامل دو میزبانه : (Shuttle vector) : حاملی که در سلولهای بیش از یک گونه بتواند همانندسازی کند ( مثلا در مخمر و E.Coli).
حوای میتوکندریایی : (Mitochondrial Eve) : زنی که در حدود 140000 تا 290000 سال قبل در آفریقا زندگی می کرده و DNA میتوکندریایی اجدادی را همراه داشته است و تمام DNAهای میتوکندریایی امروزی از آن منشا گرفته اند.
خانواده چند ژنی : (Multigene family) : تعدادی از ژن های خاص یا مرتبط موجود در یک جاندار که معمولا خانواده ای از پلی پپتیدهای مرتبط را بیان می کنند.
دئوکسیریبونوکلئاز : (Deoxyribonuclease) : آنزیمی که DNA را خرد می کند.
درز : (Nick) : شکستن پیوند و عدم وجود یک یا چند نوکلئوتید در یکی از رشته های DNA دو رشته ای.
درزگیری : (Nick translation) : ترمیم درز با DNA پلیمراز I که معمولا برای ورود کردن نوکلئوتیدهای نشاندار به درون مولکول DNA استفاده می شود .
دم گذاری هموپلیمری : (Homopplymer tailing) : اتصال توالی از نوکلئوتیدهای مشخص ( مثل AAAA) به انتهای مولکول اسید نوکلئیک. مهمولا برای ساختن هموپلیمر تک رشته ای در یک انتهای مولکول DNA دو رشته ای .
دمای ذوب (Tm) : ( Melting temperature (Tm) ) : دمایی است که در آن DNA دو رشتها ی یا هیبرید RNA-DNA دناتوره می شوند.
دناتوره شدن : (Denatureation) : در مورد مولکولهای اسید نوکلئیک ، شکست پیوندهای هیدروژنی میان جفت بازها به صورت شیمیایی یا فیزیکی.
دی دئوکسینوکلئوتید : (Dideoxynucleotide) : نوکلئوتید تغییریافتهای که گروه هیدروکسیل ‘3 ندارد و بنابر این اگر به رشته پلینوکلئوتید در حال ساخته شدن اضافه شود ، از ادامه واکنش جلوگیری میکند .
رتروویروس : (Retrovirus) : ویروسی است که ژنوم RNA دارد و می تواند به درون کروموزوم میزبان وارد شود . از مشتقات این ویروس ها می توان برای کلون کردن ژن در سلول های پستانداران استفاده کرد.
ردپانگاری : (Footprinting) : تعیین جایگاه اتصال به پروتئین روی یک مولکول DNA با تعیین این که کدام یک از پیوندهای فسفودی استری از برش با DNasel در امان مانده اند .
رزین : (Resin) : بستری برای کروماتوگرافی .
رسوب گذاری اتانولی : (Ethanol precipitation ) : رسوی گذاری مولکولهای اسید نوکلئیک در حضور اتانول و نمک که روشی برای تلغیظ DNA است .
روش تکثیر سریع انتهاهای cDNA : (RACE) (rapid amplification of cDNA ends(RACE)) : روش مبتنی بر PCR برای نقشه برداری انتهای مولکول های RNA
روش کلون کانتیگ : (Clone contig approach) : روشی برای توالی یابی ژنوم که در آن مولکول مورد نظر برای توالی یابی را به قطعاتی به طول چند صد تا چند میلیون باز می شکنند و جداگانه توالی یابی می کنند .
رونوشتبردار معکوس : (Reverse transcriptase) : DNA پلیمراز وابسته به RNA که می تواند مولکول DNA مکمل را از روی الگویی که RNA تک رشته ای است ، بسازد .
رویکرد ضربتی : (Shotgun approach) : راهکارهای برای توالییابی ژنوم که در آن مولکول هایی که باید توالی یابی شوند ، به طور تصادفی به قطعاتی بریده شده و جداگانه توالی یابی می شوند.
ریبونوکلئاز : (Ribonuclease) : آنزیمی که RNA را هضم می کند .
ریزآرایه : (Microarray) : مجموعهای از cDNAهای تثبیت شده روی ورقه شیشه ای که در مطالعات ترنسکریپتوم استفاده می شوند .
ریزتزریق : (Microinjection) : روشی برای وارد کردن DNA به درون سلول با تزریق مستقیم به درون هسته .
ژن کاندیدا : (Candidate gene) : ژنی که از طریق کلون کردن جایگاهی شناسایی شده و احتمالا عامل ایجاد یک بیماری است .
ژن گزارشگر : (Reporter gene) : ژنی که فنوتیپ آن را می توان در موجود زنده ترانسفورم شده اندازه گیری کرد . از این روش برای آنالیز حذف مناطق کنترل کننده ژن ها استفاده می کنند .
ژل الکتروفورز : (Gel electrophoresis) : الکتروفورزی که در یک زمینه ژلی انجام می شود بنابراین مولکول های با بار الکتریکی مشابه را می توان بر اساس اندازه از هم جدا کرد .
ژن : (Gene) : بخشی از DNA که یک RNA یا پلیپپتید را کد می کند.
ژن درمانی : (Gene therapy ) : فرایند بالینی که از ژن یا دیگر توالی های DNA برای درمان بیماری استفاده می کند.
ژنتیک : (Genetic) : شاخ های از زیست شناسی که به مطالعه ژن ها می پردازد.
ژنوم : (Genome ) : مجموعه کامل ژن های یک موجود زنده.
ژنومیکس : (Genimics) : مطالعه یک ژنوم ، بویژه توالی کامل یک ژنوم.
ژنومیکس عملکردی : (Functional genomics) : مطالعاتی با هدف مشخص کردن تمام ژن های ژنوم و تعیین عملکرد آنها.
ساخت مصنوعی ژن ها : (Artificial gene synthesis) : ساختن یک ژن مصنوعی با استفاده از مجموعه ای از الیگونوکلئوتیدهایی که با هم همپوشانی دارند .
سازگاردهنده : ( Adaptor) : الیگونوکلئوتید مصنوعی دو رشتهای که برای متصل کردن انتهاهای چسبنده به انتهاهای صاف (تراز) استفاده میشود .
سازگاری : (Compatibility) : امکان حضور همزمان دو نوع پلاسمید در یک سلول .
ساعت مولکولی : (Molecular clock) : روش آنالیز بر مبنای سرعت موتاسیون که امکان تعیین زمان لازم برای رسیدن به شاخه های یک درخت ژنی را مشخص میکند .
سانتریفوژ شیب چگالی : (Density gradient centrifugation ) : جداسازی مولکول ها و ذرات بر اساس چگالی شناوری با سانتریفوز کردن در محلول غلیظ ساکارز یا کلری سزیم .
ستون چرخشی : (Spin column) : روشی برای تسریع کروماتوگرافی تعویض یونی با سانتریفوژ کردن ستون کروماتوگرای.
سرکوب : (Repression) : خاموش شدن بیان یک یا چند ژن در پاسخ به یک ماده شیمیایی یا دیگر تحریکات.
سکوناز : (Sequenase) : آنزیمی که برای توالی یابی DNA با روش خاتمه سنتز استفاده می شود.
سلول بنیادی جنینی : (Embryonic stem cell(ES)) : سلولهای بنیادی حاصل از جنین موش یا موجودات زنده دیگر که برای ایجاد جانوران ترانس ژنیک، مثل موش ناکاوت شده ، استفاده می شوند.
سونیکاسیون : ( Sonication ) : فرآیندی که از فراصوت برای ایجاد شکست های تصادفی در مولکولهای DNA استفاده می شود.
سیستم ترجمه عاری ا زسلول : (Cell-free translation system) : عصاره سلولی که تمام اجزلی لازم برای ساختن پروتئین ( از جمله زیر واحدهای ریبوزومی، Trna ها ، آمینواسیدها ، آنزیمها و کوفاکتورها )) را دارد و می تواند مولکول های rna اضافه شده به مجموعه را ترجمه کند.
سیستم هیبرید دوگانه مخمر : (Yeast two hybrid system) : از روش های کلون کردن در ساکارومیسس سرویزیه برای شناسایی پروتئین هایی که با یکدیگر برهم کنش دارند .
شاخص انتخاب : (Selectable marker) : ژنی که روی حامل است و امکان شناسایی سلولی را که حامل یا DNA نوترکیب مشتق از حامل را دارد ، میسر میسازد .
شستشو : (Elution) : جدا کردن مولکول از ستون کروماتوگرافی .
طیف سنجی جذب UV : (UV absorbance spectrophotometry) : روشی برای اندازه گیری غلظت یک ترکیب یا مشخص کردن مقدار نور UV جذب شده توسط نمونهای از آن .
عصاره سلولی : (Cell extract) : فرآرده ای که محتوی تعداد زیادی از محتویات سلول های شکسته شده است .
عصاره شفاف شده : (Cleared lysate) : عصاره سلولی سانتریفوژ شده ای که قطعات سلولی، اندامک و احتمالا DNA کروموزومی از آن جدا شده است.
عنصرP : (P element) : ترانسپوزون دروزوفیلا ملانوگاستر که به عنوان پای های برای حامل کلون سازی در این حشره استفاده می شود.
غربالگری ایمونولوژیک : (Immunoligical screening) : استفاده از یک آنتی بادی برای شناسایی پلیپپتیدی که توسط یک ژن کلون شده تولید می شود.
غربالگری ترکیبی : (Combinatorial screening) : روشی که در آن با مخلوط کردن نمونه ها با نظم و ترتیب خاص می توان تعداد دفعات PCR یا آنالیزهای دیگر را کاهش داد ، بنابراین حتی بدون آزمایش کردن اختصاصی نمونه می توان آن ها را ارزیابی کرد.
غیر فعال سازی ناشی از وارد شدن : (Insertional inactivation) : یکی از راهکارهای کلون کردن که وارد شدن یک قطعه DNA به درون حامل موجب غیر فعال شدن ژن درون حامل می شود.
فارمینگ : ( Pharming ) : تغییرات ژنتیکی حیوانات اهلی به طوری که بتوانند پروتئین های دارویی نوترکیب را در شیرشان تولید کنند .
فاژکمک کننده : (Helper phage) : فاژی که به همراه حامل کلون سازی مربوطه اش به سلول میزبان وارد می شود تا آنزیم های لازم برای همانند سازی حامل کلون کردن را فراهم کند .
فاژمید : (Phagemid) : پلاسمید دو رشته ای که منشا همانندسازی یک فاژ رشته ای را دارد و می توان از آن برای تهیه نمونه تک رشته ای ژن کلون شده استفاده کرد.
فراوانی ترانسفورم کردن : ( Tranformation frequency) : تعداد سلول هایی که در یک آزمون ترانسفورم شده اند.
فن آوری آنتی سنس : (Antisense technology) : استفاده از زن کد کننده RNA آنتی سنس در مهندسی ژنتیک.
فنآوری خاتمهگر : (Terminator technology) : فرایند نوترکیبی DNA که موجب ساخته شدن پروتئین مهار کننده ریبوزوم در جنین گیاهان می شود و برای جلوگیری از امکان کاشت دانه گیاهان مهندسی یشده استفاده می شود.
فنآوری DNA نوترکیب : (Recombinant DNA technology) : روش هایی برای ساختن، مطالعه و استفاده از مولکول های DNA نوترکیب.
قطعه کلنو ( از DNA پلیمراز I ) : (Klenow fragment (of DNA polymerase I)) : آنزیم DNA پلیمرازی است که از ایجاد تغییرات شیمیایی در DNA پلیمراز I اشرشیا کلی حاصل می شود و در توالی یابی DNA کاربرد دارد.
قوانین واتسون و کریک : ( Watson – Crik rules) : قوانین جفت شدن بازها که در ساختار ژن ها و بیان آنها صادق است . طبق این قانون A با T و G با C جفت میشوند .
کاست : (Cassette) : توالی DNA مشتمل بر پروموتر ، جایگاه اتصال ریبوزوم ، جایگاه برش آنزیمهای محدودگر ، ترمیناتور( یا برای میزبان های یوکاریوتی، پروموتر ، جایگاه های برش آنزیم های محدودگر، توالی پلیآدنیلاسیون) که درون حامل بیانی وجود دارد. اگر ژن خارجی درون جایگاه های برش با آنزیم های محدودگر وارد شود تحت کنترل علایم بیانی حامل قرار گرته و بیان می شود.
کاستن ژنی : (Gene subtraction) : یک راهکار مهندسی ژنتیک که موجب غیر فعال شدن یک یا چند ژن یک موجود زنده می شود .
کاسمید : (Cosmid) : حامل کلون سازی پلاسمیدی که جایگاه COS فاژ λ را دارد. از این حامل ها برای کلون کردن قطعات DNA تا kb 40 استفاده می شود .
کایمر : (Chimera) : (1) مولکول DNA نوترکیبی که از قطعات DNA چند موجود زنده تشکیل شده است . این اسم از نام جانوری افسانهای گرفته شده است . (2) محصول اولیه کلون کردن با استفاده از سلول های بنیادی جنینی : جانوری که از مجموعه ای از سلول ها با ژنوتیپ متفاوت تشکیل شده است .
کانتینگ : (Contig) : یک قطعه پیوسته از توالی DNA ، حاصل بخشی از یک پروژه توالییابی ژنوم .
کپسید : (Capsid) : پوشش پروتئینی که DNA یا RNA باکتریوفاژ یا ویروس را می پوشاند .
کتابخانه ژنومی : ( Genimic library) : مجموعه ای از کلونهایی که کلیه ژنهای موجود زنده را دارد .
کتابخانه نمایش فاژی : (Phage display library ) : مجموعه ای از کلون های M13 که قطعه متفاوتی از DNA مورد استفاده در نمایش فاژی را همراه دارند .
کروماتوگرافی تعویض یونی : (Iin exchange chromatography ) : روشی برای جداسازی مولکول ها با توجه به قدرت اتصال آن ها به ذرات باردار الکتریکی موجود در یک ماتریکس کروماتوگرافی .
کروماتوگرافی تمایلی : (Affinity chromatography) : روش کروماتوگرافی مبتنی بر استفاده از یک لیگاند ه به پروتیین خاصی متصل می شود و بنابراین میتوان از آن برای خالص سازی پروتئین مورد نظر استفاده کرد .
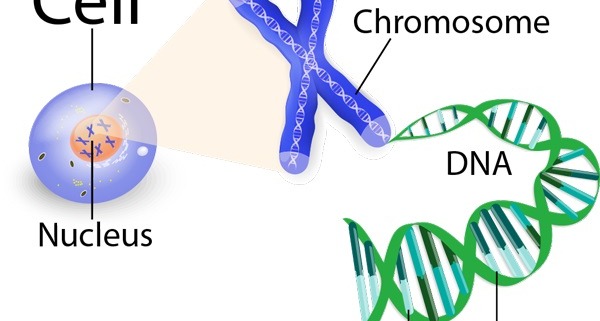
کروموزوم : (Chomosome ) : ساختاری متشکل از DNA و پروتئین که بخشی از ژنوم هسته ای یوکارت ها را در بر دارد. با کمی تسامح می توان از این اسم در مورد ژنوم پروکاریون ها نیز استفاده کرد .
کروموزوم مصنوعی باکتریایی (BAC) : (Bacterial artificial chormosome) : حامل کلون سازی با پایه پلاسمید F که برای کلون کردن قطعات بزرگ DNA در اشرشیا کلی استفاده می شود.
کروموزوم مصنوعی مخمر : (Yeast artificial chormosome (YAC)) : حامل کلون سازی که ترکیب ساختاری کروموزوم مخمر را دارد و از آن می توان برای کلون کردن قطعات بزرگ DNA استفاده کرد .
کروموزوم مصنوعی مشتق شده از P1)) (PAC) : حامل کلون کردن با پایه باکتریوفاژ P1 که برای کلونسازی قطعات بزرگ DNA در اشرشیا کلی استفاده میش.د .
کروموزوم ویروس : (Virus chromosome ) : مولکول های RNA یا DNA که درون یک کپسید ویروسی هستند و ژن های ویروسی را حمل می کنند.
کشت بسته : (Batch culture) : کشت باکتری ها در حجم ثابتی از محیط کشت مایع ، در یک ظرف بسته را می گویند. در این روش در طی مدت گرماگذاری ، اضافه کردن یا کم کردن محیط کشت انجام نمی شود.
کشت مداوم : (Continuous culture) : کشت میکروارگانیسم ها در محیط کشت مایع تحت شرایط کنترل شده همراه با اضافه کردن و برداشت محیط کشت در دامنه های زمانی خاص.
کشت مایع : (Broth culture) : کشت میکروارگانیسم ها در محیط کشت مایع.
کلون : (Clone ) : مجموعه ای از سلول ها که مولکول های DNA نو ترکیب یکسانی دارند.
کلون کرد DNA مکمل (cDNA) : ( co,plemntary DNA (cDNA) cloning ) : روش کلون کردن مبتنی بر استفاده از DNA از روی mRNA .
کلون کردن جایگاهی : (Position cloning ) : فرآندی که از اطلاعات جایگاه یک ژن برای بدست آوردن کلون آنژن استفاده می شود.
کلون کردن ژن : (Gene cloning ) : وارد کردن قطعه DNAای که حامل یک ژن است به درون یک حامل کلون سازی و تکثیر مولکول DNA نوترکیب حاصل در یک میزبان ، از این اصطلاح برای توصیف روش های هوشمندانه ای که بدون حامل کلون سازی موجب انتقال ژن می شوند ( مثل روش انتقال مستقیم ژن ) هم استفاده می شود.
کلون کردن ضربتی : (Shotgun cloning) : راهکاری برای کلون کردن که شامل وارد کردن قطعات تصادفی یک مولکول DNA بزرگ به درون حامل است که منجر به ایجاد تعداد زیادی مولکول DNA نوترکیب می شود.
گروه ناسازگار : ( Incompatibility group ) : انواع مختلفی از پلاسمیدها که اغلب از یک خانواده هستند ، اما نم یتوانند به همراه هم در یک سلول وجود داشته باشند.
گام زنی کروموزومی : ( Choromosome walking) : روشی برای ایجاد کلون کاتینگ با شناسایی قطعات DNA کلون شده ای که با هم همپوشانی دارند.
گسترش پرایمر : ( Primer extension ) : روشی برای تحلیل رو نوشت که در آن انتهای’5 یک RNA با اتصال و گسترش یک پرایمر الیگونوکلئوتیدی نقشه برداری میشود .
لامبدا : ( Lambda) ( λ ) : باکتروفاژی که اشرشیا کلی را عفونی می کند. از مشتقات آن به عنوان حامل کلون کردن استفاده می شود.
لیزوزیم : ( Lysozyme) : آنزیمی که دیواره سلولی برخی باکتریها را تضعیف می کند.
لیگاز (DNA لیگاز ) : (Ligase (DNA ligase) : آنزیمی است که بریدگیهای مولکولهای DNA دو رشته ای را در سلول ها ترمیم می کند . در آزمایش های کلون سازی از DNA لیگاز خالص شده برای متصل کردن مولکول های DNA استفاده می شود.
مبدا همانندسازی : (Origin of replication ) : جایگاه مخصوصی روی مولکول DNA که همانندسازی DNA از آنجا شروع می شود.
متصل کننده : ( Linker ) : یک الیگونوکلئوتید دو رشته ای مصنوعی است که برای اتصال انتهاهای چسبنده به مولکول های با انتهای صاف استفاده میشود .
محدودسازی بیولوژیکی : (Biological containment) : عبارت از روش های پیشگیرانه ای است که برای جلوگیری از تکثیر مولکول های DNA نوترکیب در میکروارگانیسم ها در محیط طبیعی انجام می شود. در این روش از حامل ها و میزبان هایی استفاده می شود که دستکاری شده اند و بنابراین نمی توانند خارج از محیط آزمایشگاه زنده بمانند .
محیط کشت حداقل : ( Minimal medium ) : محیط کشت مشخصی که تنها تعداد محدودی از مواد غذایی مورد نیاز برای یک باکتری خاص را دارد .
محیط کشت مشخص : ) Defined medium ) : محیط کشت باکتریایی که ترکیباتش معلوم است .
محیط کشت نامشخص : ( Undefined medium ) محیط کشتی که اجزای تشکیل دهنده اش مشخص نیستند.
مکمل : ( Complementary) : دو پلینوکلئوتیدی که بازهایشان می توانند با هم جفت شوند و یک مولکول دو رشته ای ایجاد کنند.
مولکول DNA نوترکیب : ( Recombinant DNA molecule) : مولکول DNA ای که با متصل کردن قطعات DNA مختلف به یکدیگر به دست می آید.
معرف نقشه برداری : ( Mapping reagent ) : مجموعه ای از قطعات DNA روی کروموزوم ها یا درون ژنوم که برای نقشه برداری STS استفاده می شوند.
موش ناکاوت شده : ( Knockout mouse ) : موشی که مهندسی شده و حامل یک ژن غیر فعال است .
مولد : (Productive ) : چرخه عفونی ویروس که می تواند به تکمیل ساخته شدن و رها شدن ذرات ویروسی جدی بی انجامد .
مهندسی پروتئین : (Protein engineering) : مجموعه روش هاییکه شامل ایجاد جهش در ژ«ن ها و ایجاد تغییرات هدفمند در مولکول پروتئین سنتز شده است . اغلب از این روش ها برای بهبود ویژگیه ای آنزیم هایی که کاربرد صنعتی دارند استفاده می شود.
مهندسی ژنتیک : (Gnetic engineering ) : استفاده از روش های آزمایشگاهی برای ایجاد مولکول های DNA حاوی ژن یا ژن های جدید.
میدان الکتریکی یکنواخت حاشی های : ( Contour clamped ho,ogeneous elecric fields (CHEF) ) : روش الکتروفورز برای جدا کردن قطعات بزرگ DNA .
نشاندار کردن : ( Labelling ) : وارد کردن یک نوکلئوتید نشانگر به درون مولکول اسید نوکلئیک . نوکلئوتید نشانگر معمولا ( و نه همیشه ) رادیواکتیو یا فلورسنت است .
نشانگر DNA : ( DNA marker ) : توالی DNAکه به صورت دو یا چند الل وجود دارد و بنابراین می توان از آن در نقشه برداری ژنتیکی استفاده کرد.
نشانگر رادیواکتیو : ( Radioactive marker) : اتم رادیو اکتیو مورد استفاده برای شناسایی مولکول بزرگی که درون آن وارد شده است .
نقشه برداری ژنی : (Gene mapping ) : تعیین جایگاه نسبی ژن های مختلف روی یک مولکول DNA .
نقسه برش با آنزیمهای محدودگر : (Restriction map) : نقش های که نشان دهنده جایگاه برش آنزیم های محدودگر مختلف روی مولکول DNA است .
نقشه ژنتیکی : ( Genetic map ) : نقشه ژنومی که از بررسی مستقیم مولکول های DNA حاصل شده است .
نمایش فاژی : ( phage display) : روشی مبتنی بر لون کردن در فاژ M13 ، برای شناسایی پروتئین هایی که با یکدیگر برهم کنش دارند .
نوترکیب : ( Recombinant) : سلول ترانسفورم شده ای که دارای یک مولکول DNA نوترکیب است .
نوترکیبی : (Recombination) : تعویض توالی های DNA میان دو مولکول مختلف . نوترکیبی به طور طبیعی رخ می دهد اما در دستکاری های DNA هم از آن استفاده می شود.
نوترکیبی همولوگ : ( Homologous recombination ) : نو ترکیبی میان دو مولکول DNA دو رشته ای همولوگ، از جمله آن هایی که تشابه توالی نوکلئوتیدی گسترده ای دارند.
واکنش زنجیرهای پلیمراز : ( Polymerase chain reactoin(PCR)) : روشی برای تکثیر آنزیمی نسخه های متعدد از توالی DNA هدف .
وکتور یا حامل : ( vector) : مولکول DNA که می تواند در موجود زنده میزبان تکثیر یابد. برای کلون کردن ، ژن را وارد آن کرده و مولکول DNA نوترکیب را به دست میآورند .
ویروس کلم (Caulimoviruses) : یک از دو گروه ویروس DNA داری که گیاهان را عفونی میکنند . این ویروس ها پتانسیل لازم برای استفاده شدن به عنوان حامل کلون کردن ژن در تعدادی از گیاهان عالی را دارند .
ویروس موزائیک کلم : ( cauliflower mosaic virus (CaMV)) : شناخته شده ترین ویروس کلم که در گذشته به عنوان حامل کلون کردن برخی گونه های گیاهی عالی استفاده میشد . ویروس موزائیک کلم منبعی برای تهیه پروموترهای قدرتمند در انواع دیگر حامل های کلون سازی گیاهی نیز می باشد.
ویروس همراه آدنو (AAV) : (Adeno-associated virus (AAV)) : ویروسی که ارتباطی با آدنوویروس ها ندارد ، اما اغلب در بافت های عفونی شده با آدنوویروس ها دیده می شود، زیرا AAVها از برخی پروتئین های تولید شده توسط آدنوویروس ها برای تکمیل چرخه همانند سازی خود استفاده می کنند.
هاپلو گروه: ( Haplogroup) : یکی از کلاس های اصلی توالی DNA میتوکندری موجود در جمعیت انسانی .
هضم دوگانه : (Double digestion) : برش مولکول DNA با دو آنزیم محدودگر مختلف ، همزمان یا پشت سرهم .
هضم ناقص : ( Partial digestion ) : تیمار مولکول DNA با یک آنزیم محدودگر تحت شرایطی که تنها بخشی از جایگاه های برش توسط آنزیم شناسایی و بریده شوند .
هم یوغی : ( Conjugation ) : اتصال فیزیکی میان دو باکتری که معمولا همراه با انتقال DNA از یک سلول به سلول دیگر است .
همولوژی (Homology) : به دو ژن حاصل از دو موجود زنده مختلف گفته می شود که از یک ژن اجدادی مشتق شده اند. دو ژن همولوگ معمولا تشابه توالی لازم برای فراهم کردن امکان استفاده از یکی به عنوان پروب شناسایی دیگری را دارند .
هیبریداسیون اسیدنوکلئیک : ( Nucleic acid hybridization ) : ایجاد یک مولکول دو رشته ای در اثر جفت شدن بازهای پلی نوکلئوتیدهای مکمل یا همولوگ .
هیبریداسیون در محل : ( In situ hybridization) : روشی برای نقشه برداری ژن که شامل هیبریداسیون یک نمونه نشاندار از ژن کلون شده با مولکول بزرگی از DNA ( که معمولا کروموزوم است ) می باشد.
هیبریداسیون در محل فلورسنسی : ) Fluoresccence in situ hbridization) : یک روش هیبریداسیون در محل که از فلوروکرومهایی با رنگ های مختلف برای جایابی دو یا چند ژن روی کروموزوم تحت آزمایش استفاده می شود.
DNA باستانی : (Ancient DNA) : DNA حفظ شده در یک نمونه باستان شناسی یا فسیل .
DNA پلیمراز : (DNA polymearase) : آنزیمی که از روی الگوی DNA یا RNA ، DNA می سازد.
DNA تام سلول : (Total cell DNA) : متشکل از کل DNA موجود در یک سلول یا در گروهی از سلول ها.
DNA پلیمراز Taq : (Taq DNA polymerase) : DNA پلیمراز مقاوم به حرارت که در PCR استفاده می شود.
DNA حلقوی باز : ((OcDNA) Open-circular DNA) : آرایش فضایی غیر ابرمارپیچی DNA که در هنگام وجود درز در DNA حلقوی دو رشته ای ایجاد می شود.
DNA حلقوی بسته کووالان : ( Covalently closed-circular DNA (cccDNA)) : مولکول DNA حلقوی دو رشته ای بدون درزی که معمولا به صورت ابرمارپیچ دیده می شود.
DNA ژنومی : (Genomic DNA) : متشکل از کل DNA موجود در یک سلول یا گروهی از سلول ها.
DNA PCR تکراری : ( Repetitive DNA PCR ) : روش انگشت نگاری کلون که از PCR برای مشخص کردن جایگاه های مربوط به توالی های تکراری دورن قطعات DNA کلون شده استفاده می شود.
GM ( محصولات مهندسی شده ) : (GM ( Genetically modified ) crop ) : گیاهی که با اضافه کردن یا کاستن ژنها حاصل شده است.
Heterologous probing : استفاده از یک مولکول اسید نوکلئیک نشاندار برای مشخص کردن مولکوله ای مشابه با روش هیبریدسازی.
Host controlled restriction : مکانیسمی است که بوسیاه آن برخی باکتری ها از حمله فاژها جلوگیری می کنند. آن ها این کار را با تولید اندونوکلئازهای محدودگری که DNA غیر باکتریایی را می شکنند، انجام می دهند.
Indel : جایگاهی که یک توالی DNA وارد ژنوم می شود یا از ژنوم خارج می شود. این نام گذاری به خاطر این است که با مقایسه دو توالی جایگزین نمی توان گفت که آیا توالی از یک حذف شده یا به دیگری اضافه شده است.
M13 : باکتریوفاژی است که اشرشیا کلی را آلوده می کند. از مشتقات آن به عنوان حامل کلون سازی استفاده می شود .
M13 تکثیری : شکل دو رشتهای مولکول M13 DNA که در سلول های اشرشیا کلی آلوده شده دیده می شود.
Orphan : ORFای که به نظر میرسد مربوط به یک ژن عملکردی باشد ، اما هیچ عملکردی از آن مشاهده نشده باشد.
ماهوارک : (Microsatellite) : پلیمورفیمس است که معمولا شامل تکرار توالی های دو ، سه ، چهار یا پنج نوکلئوتیدی است. به این توالی ها توالی های کوتاه تکراری پشت سر هم نیز گفته می شود (STR) .
P1 : باکتریوفاژی که اشرشیا کلی را عفونی می کند و از مشتقات آن به عنوان حامل کلون سازی استفاده می شود .
Principle Component analysis : فرایندی برای تعیین الگوی کلی در مجموعه بزرگی از نمونه های متفاوت .
Processivity : مقدار DNAای که قبل از جدا شدن DNA پلیمراز از الگو ساخته می شود .
Radiation hybrid : مجموعه های از رده های سلولی جندگان که دارای قطعات مختلف ژنوم انسان هستند و با روش پرتودهی ساخته شده اند . از این مجموعه به عنوان وسیله نقشه برداری برای مطالعه ژنوم انسان استفاده می شود .
Random priming : روشی برای نشاندار کردن DNAبا استفاده از هگزامرهای تصادفی که به صورت تصادفی به DNA تک رشته های متصل شده و نقش پرایمر را برای ساختن رشته مکمل الگو توسط آنزیم مناسب بازی می کنند .
Relaxed : DNA حلقوی بدون ابر مارپیچ .
RNA پیامبر : رو نوشت ژن های کد کننده پروتئین .
PCR چندگانه : (Multiplex PCR ) : PCRای که با بیش از یک جفت پرایمر انجام میشود و دو یا چند جایگاه را روی قطعه DNA مورد نظر هدف گیری می کند.
PCR قطعه تکراری پراکنده : ( IRE-PCR) : یک روش انگشت نگاری کلون با استفاده از PCR برای شناسایی جایگاه نسبی توالی های تکراری در قطعات DNA کلون شده.
RT-PCR : روش PCR که در آن ماده اولیه شروع واکنش RNA است . در مرحله اول این فرایند با واکنش رونوشت برداری معکوس یک مولکول c DNA از RNA ، ساخته می شود.
Simian virus 4 (SV40) : ویروس پستانداران که به عنوان پایه حامل های کلون سازی استفاده شده است .
Stem-Loop : ساختار سنجاق سری پلینوکلئوتیدها که دارای ساقه ای حاصل از جفت شده بازها و یک لوپ جفت نشده است.
Stuffer fragment : بخشی از حامل جایگزینی λ که برای ورود DNA جدید از آن حذف می شود .
T-DNA : بخشی از پلاسمید Ti که وارد DNA گیاه می شود.
Vehicle یا حامل : کلمه ای که گاه به جای وکتور به کار می رود. معنی آن این است که حامل ، ژن کلون شده را در طی فرایند کلون کردن انتقال می دهد.
Wave of advance model : تئوری که بر اساس آن گسترش کشاورزی در اروپا با حرکت گسترده انسان همراه شده است .
Whole genome shotgun approach : راهکار توالییابی ژنوم که ترکیب توالی یابی ضربتی تصادفی با یک نقشه ژنومی است. در اینجا از نقشه ژنومی برای سر هم کردن توالی کلی استفاده می شود.
Zoo blot : غشای نایلونی یا نیتروسلولزی که DNA تثبیت شده چندین گونه را دارد و برای مشخص کردن وجود یا عدم وجود همولوژی بین یک ژن از یک گونه با ژن هایی از گونه های دیگر استفاده می شود.
گردآورنده : سرکار خانم پریناز زارع
رفرنس: کتاب کلون سازی ژن ها و آنالیز DNA نوشته پروفسور براون








بسیار عالی، سپاس
ممنون از همراهی شما